Welcome to WordPress.com. This is your first post. Edit or delete it and start blogging!
El "toma y daca" en la simbiosis legumbre-rizobio

Las plantas requieren nitrógeno para la generación de compuestor orgánicos. Sin embargo en la naturaleza la mayoría del nitrógeno se encuentra como N2, una molécula muy inerte. Para poder aprovecharlo las plantas se asocian a bacterias, las cuales son capaces de generar amonio (NH4) a partir del nitrógeno gaseoso.
En legumbres uno de los fijadores de nitrógeno por excelencia son bacterias del genero Rhizobium (del griego, que vive en la raíz) como por ejemplo Rhizobium leguminusarum. Esta bacteria infecta las raíces, formando nódulos en las raíces de las plantas donde gracias al ambiente anaerobio, son capaces de fijar nitrógeno. En el proceso de infección muchas cosas le ocurren a la bacteria: se diferencia a un bacteroide con una morfología claramente diferente entre otras cosas.
Ahora bien, ¿qué gana la bacteria con esto? Se sabe que la bacteria «da» amonio a la planta pero además recibe «a cambio» ácidos dicarboxílicos como fuente de carbono. Sin embargo en este bonito trabajo publicada en PNAS se demuestra que el intercambio es un poco más complejo y que existen algunos cambios sorprendentes en la bacteria.
Se sabía que aún si no se tocaban los genes de fijación de nitrógeno o los genes de importación de ácidos dicarboxílicos algunos transportadores de la familia ABC seguían siendo esenciales en el establecimiento de la relación simbiótica. Por experimentos genéticos se dan cuenta que estos transportadores son específicos de leu, ileu y val. Por otro lado obtienen por microarray y qPCR que los genes de la biosíntesis de estos aminoácidos…se apagan!! Es decir que bacterias que son protótrofas para esos aminoácidos se hacen auxotrofas en condiciones de simbiosis. Los autores llaman a este fenómeno «auxotrofía simbiótica». Al parecer el suministro de estos aminoácidos a la bacteria por parte de la planta es crítica en el establecimiento de la relación simbiótica y su persistencia!
5to Microvermú
Como hemos hecho desde abril de este año los últimos viernes de cada mes nos reunimos nuevamente para escuchar y aprender de otr@s microbiólogos. Esta vez nos contarán cómo hacer pilas con ….bacterias!!!
25 de septiembre — 18:30 hs — 5to Microvermú
Aula Cardini, Dtpo. Química Biológica, Facultad de Ciencias Exactas y Naturales- UBA, pabellón II Ciudad Universitaria
Nos habla Natalia Sacco sobre…
«Caracterización de Cepas Microbianas Adecuadas para ser Utilizada en Celdas de Combustible Microbianas (MFCs) de Tercera Generación. Aplicaciones como Transductores para la Medición de Parámetros de Calidad de Aguas»
La conversión de energía química en eléctrica es posible en dispositivos electroquímicos denominados pilas de combustible (fuel cells), donde la electricidad se obtiene a partir de una fuente externa de combustible química que suele ser hidrogeno; sin embargo, la fuente primaria para la producción de H2 sigue siendo principalmente los combustibles fósiles.
El principio operacional de las MFCs se basa en la extracción y transferencia de electrones desde el metabolismo microbiano (cadena de transporte de electrones) hasta el ánodo. El ánodo es conectado a través de una resistencia de carga (RL) al cátodo, vía un circuito eléctrico externo, a través del cual fluyen los electrones, originando una corriente (I). Los electrones viajan del ánodo (negativo) al cátodo (positivo) debido a la diferencia de potencial rédox que existe entre las soluciones catódica y anódica. Se ha demostrado que la transferencia directa de electrones desde las células microbianas hacia los electrodos ocurre con muy baja eficiencia, a menos que bacterias de algunos grupos especiales (Geobacter y otras), sean utilizadas.
Las MFCs se han clasificado en tres grupos: las denominadas de primera generación, donde mediadores rédox artificiales (rojo neutro, azul de metileno, etc.) son capaces de atravesar la membrana citoplasmática celular y transferir electrones desde éstas hacia la superficie del ánodo. En las de segunda generación, los electrones son transferidos mediante la reducción y oxidación de compuestos de azufre, mientras que las de tercera generación utilizan bacterias reductoras de metales (miembros de la familia Geobacteraceae y Shewanellaceae) que presentan citocromos unidos a membrana, capaces de transferir electrones directamente a los electrodos. Existe también la posibilidad de alojar una pila de combustible en un hábitat natural o de materiales recolectados, y obtener así energía eléctrica a partir de las comunidades microbianas indígenas. En este caso, el diseño recibe el nombre de “celda de combustible sedimentaria” y requiere el contacto del ánodo en el sedimento anaerobio que hace las veces de cámara anódica, mientras que el cátodo queda expuesto en la fase acuosa aeróbica que cubre el sedimento.
Las MFCs han sido utilizadas principalmente como métodos de producción de energía eléctrica para aplicaciones de muy baja potencia, en las que estos sistemas pueden tener ventajas respecto a las fuentes de poder convencionales. Nosotros en esta tesis proponemos utilizarlas como transductores del metabolismo microbiano. Varios parámetros bioanalíticos, tales como BOD y toxicidad están relacionados a la actividad metabólica microbiana, o a los cambios producidos por esta actividad microbiana en los medios de cultivos o soluciones que contienen estos microbios.
Los esperamos! Habrá Cinzano y papitas.
Publicado en Uncategorized
Localización de proteínas en bacterias mediada por curvatura!
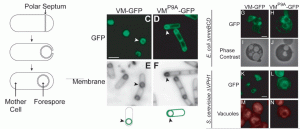
Cada vez más se esta dejando de ver a la bacteria como una bolsa de protoplasma con poco más que ADN y ribosomas aislados. Siempre se enseña y se dice que las bacterias no tienen histonas, ni citoesqueleto, ni organielas, etc. Sin embargo de cierta manera también existen con sus variaciones. Si bien el trafico proteico no está tan bien caracterizado ni es tan conspicuo como en Eucarya sí existe y siempre determinadas proteínas van a un determinado sitio en la célula bacteriana. Ejemplos claros son las proteínas de la maquinaria de competencia en B.subtilis que se localizan en uno de los polos o las proteínas del septo que van al centro de la célula a punto de dividirse. Acá les acerco un trabajo buenísimo de Richard Losick que me partio bastante la cabeza. Trabajan con la localización de SpoVM (VM) una proteína que localiza en la espora de Bacillus subtilis. En las esporulación, B.subtilis se divide asimétricamente y luego la célula madre incluye a la futura espora. VM se localiza justamente rodeando la espora pequeña, y sólamente ahí. Como lo consigue no se sabía. Sí se sabía que VM es una alfa hélice anfipática que se asocia a la membrana a lo largo de su eje largo. Además la mutacion de una prolina (sí, justo una prolina!! ya verán mas adelante!)en posicion 9 por una alanina generan una localizacion generalizada a cualquier membrana (VMp9). Se preguntaron qué es lo que genera esa localización. Encontraron que la curvatura positiva en la membrana de la espora generaba estas diferencias. Mutantes con una membrana con protrusiones internas generaba la localización de VM-GFP pero no VMp9-GFP en dichos sitios. Pero no se limitaron a eso, produjeron un vector que expreaba la proteina de manera inducible. Luego introdujeron el vector en cepas mutantes de E.coli o de levadura que generaban vesículas internas. Al inducir la expresión de VM-GFP la proteína se localizaba en la superficie de las vesículas internas!! La mutante VMp9-GFP se asociaba a todas las membranas. Además también lo hacen en vesículas desnudas con el mismo resultado. Concluyen que lo que guía la localización de esta proteína es exlusivamente la curvatura de la membrana (y su grado de curvatura)!!
Publicado en bacillus, espora, localizacion de proteinas
Anti-anti sense en el sistema toxina-antitoxina !! Un poco de Biotecnologia…
Un lindo trabajo de Liam Good y su grupo un investigador sueco que viene haciendo muy buen trabajo en el campo de la tecnología antisentido en sistemas procariotas. Su grupo ha desarrollado a lo largo de estos años oligos de análogos de acidos nucleicos resistentes a nucleasas: PNAS (peptide nucleic acids) que son basicamente bases de ADN unidas entre si por enlaces peptídicos.
Entre los blancos a silenciar han empleado genes vitales con el objetivo de generar nuevos antibióticos. En este trabajo sin embargo hacen algo muy elegante: silencian al represor de un sistema toxina-antitoxina. Los sitemas toxina-antitoxina estabilizan replicones en bacterias, se los ha llamado genes de «adicción al plásmido» (je! no al lexotanil, para el que tendrían receptor). Ellos trabajan con el sistema hok/sok del plásmido R1. Consiste en un gen que codifica para una toxina, hok (por host killing) y un marco de lectura superpuesto a hok necesario para la traducción de la toxina (mok por modulation of killing). Hay un tercer actor en discordia, sok (por supression of killing), un gen que transcribe un ARN chiquito antisentido de mok e inhibe su traducción. Al inhibirse mok, hok no puede traducirse y la toxina no se genera. El mensajero de hok es mucho más estable (tiene una vida medioa de 30 minutos) mientras que sok genera un ARN regulatorio mucho más lábil de una vida de 30 segundos! Si se pierde el plásmido el mensajero de la toxina es mucho más estable (y el modulador también), esta se traduce y mata a la bacteria.
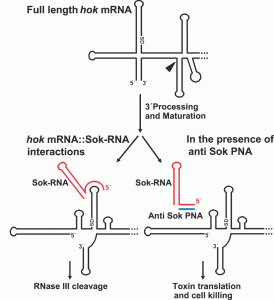
Y acá viene la excelente idea que tuvieron Good y sus muchachos: hicieron un oligo de PNA antisentido contra sok (un anti-antisense!!!). De este modo el oligo entra a la bateria y al traducirse hok, la célula muere. Pero, esto mataría sólo a la bacterias que llevan el plásmido R1? Curiosamente además en el trabajo encuentran varios homologós al sistema hok/sok en en genoma de muchas enterobacterias. Muuuy interesante.
Les dejo además una foto de un experimento muy bonito: toman células con o sin el sistema sok/hok en un plásmido. Comparan lo que pasa al meter el PNA antisene, uno control con secuencia aleatoria o rifampicina (que inhibe la transcripción).
Faridani OR, Nikravesh A, Pandey DP, Gerdes K, Good L. Competitive inhibition
of natural antisense Sok-RNA interactions activates Hok-mediated cell killing in
Escherichia coli. Nucleic Acids Res. 2006;34(20):5915-22.
Publicado en antisense, biotecnología, sistema toxina-antitoxina
4to Microvermú! Diseño de péptidos inhibitorios del Sistema de Secreción Tipo III (TTSS) de Escherichia coli y enterohemorrágico
Muchachos y muchachas! tenemos orador para la proxima microjuntada!
Nos va a hablar Mariano Larzabal, del INTA.
Recuerden que es el Viernes 28/8 a las 18hs, en el Auditorio de la FIL. Luego del stress de Conicet.
Los esperamos! Por favor difundan, habrá pizza y vermu.
Título:
Diseño de péptidos inhibitorios del Sistema de Secreción Tipo III (TTSS) de Escherichia coli enteropatogénico (E. coli enterophatogenic-EPEC) y Escherichia coli enterohemorrágico (E. coli enterohaemorrhagic- EHEC)
Objetivo:
Identificar y evaluar péptidos bloqueantes que impidan el desencadenamiento de la acción del sistema de secreción de tipo III (TTSS) y la adherencia al epitelio por parte de EHEC/EPEC.
Resultados:
Presentamos la identificación y evaluación de péptidos sintéticos como posibles inhibidores del TTSS. Tres tipos de péptidos fueron sintetizados: I) péptidos pertenecientes al domino Coiled-coil de EspA and EscF, componente mayoritario y basal del TTSS, respectivamente. II) péptido correspondiente al sitio de unión de la proteína TIR (receptor bacteriano) con la proteína intimina (adhesina bacteriana); III) péptidos seleccionados por la técnica de phage display con proteínas recombinantes EspA y EspB. Péptidos CoilA y CoilB, representan la región Coiled-coil de EspA, y CoilD, corresponde a la región Coiled-coil de EscF, produjeron inhibición de la lisis de glóbulos rojo en ensayos de hemólisis. Por lo tanto, los péptidos CoilA, B y D fueron efectivos en la inhibición del TTSS de la cepa EPEC 2348/69. Los péptidos coilA y B también mostraron ser altamente efectivos en la reducción de la formación de lesiones A/E en células HEp-2 (ensayo de FAS). Los restantes péptidos sintetizados no produjeron inhibición alguna en los ensayos realizados. Los resultados sugieren que los péptidos Coiled-coil estarían interfiriendo en el ensamble del TTSS, haciendo de estos péptidos una atractiva alternativa para reducir la patogénesis de cepas EPEC/EHEC.
Publicado en Uncategorized
Lexotanil y bacterias, tiene algo que ver ?
Como ya muchos saben, ya sea por uso personal o por uso ajeno las benzodiazepinas son utilizadas para calmar la ansiedad, desórdenes de sueño y patologías similares. Ademas del ya conocido reseptor tipo GABA el cuál es el blanco terapéutico de las benzodiazpinas, existe un segundo receptor denominado TSPO (translocator protein) ubicado en la membrana externa de las mitocondrias. El receptor TSPO se necuentra en todos los tejidos con excepción de neuronas, jugando un rol en la esteroidogénesis, sintesis de profirinas, proliferación celular y apoptosis. A pesar de la diferencias en tamaño un homólgo funcional de TSPO fue encontrado en la bacteria fotosintética Rhodobacter sphaeroides, donde regula la síntesis de pigmentos y la fotosíntesis en respuesta a la luz y las modificaciones de la presión de O2. Se encontro en otras bacterias como Flemyella diplosphon y Shinorhizobium meliloti.  Teniendo en cuenta la vida simbiotica de estas bacterias se creyo en principio que este receptor es solo un olvido evolutivo.
Teniendo en cuenta la vida simbiotica de estas bacterias se creyo en principio que este receptor es solo un olvido evolutivo.
Pero para dar un batacaso Chapalain y col. identifcaron un homólogo a tspO en distintas cepas de Pseudomonas fluorescens. (Fig 1)

demostrando in silico que existen ortólogos a TspO en eubacteria, muchas de ellas patógenas. r). No solo el tspO de Pseudomonas es funcional sino que es capaz de unir con alta afinidad el ligando específico de PK11195 del TSPO humano. Demostrado mediante un ensayo radioactivo. (Pueden ver el scatchard en la figura siguiente).
El TSPOps no solo es capaz de unir este ligando sino que al tratar las células con este ligando se afecta la capacidad de adhesión, formación de biofilm y citotoxicidad de Pseudomonas sugiriendo que el tspOps posee un importante rol en la patógenesis de Pseudomonas.
Lo que nos faltaba es que las bacterias también se vuelvan adictas a los antidepresivos.
PMID: 19564920
Publicado en biodiversidad, ecología microbiana
El fin de la bioquímica, JA, no es para tanto
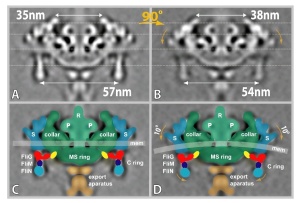
Esta es solo una nota que acabo de leer y me pareció muy impactante. Liu y colaboradores de la Universidad de Houston tomaron fotografías del motor flagelar de Borrelia bugdorferiutilizando Cryo electron tomography (Cryo–ET). OK no pienso escribir más, creo que una imagen vale mas que mil palabras.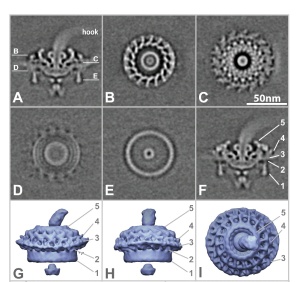

Publicado en Uncategorized
Mutaciones adaptativas
Quise postear este trabajo que me llamo la atención. Perdón por mi ignorancia pero cada vez que leo un trabajo sobre mutaciones adaptativas, siempre pienso que es un tema acabado y me termino dando cuenta que estamos igual que los primeros trabajos del tema. En mamíferos es E. coli la primer bacteria en colonizar el intestino estéril de los recién nacidos. Asi mismo la evolución de comunidades que deben adaptarse a nuevas condiciones ambientales requieren un proceso de adaptación pleiotrópica supuestamente causada por mutaciones adaptativas. En el laboratorio de Taddei, analizaron la presencia de mutaciónes adaptativas presentes en la cepa de E. coli MG1655 al colonizar el tracto digestivo (TI) de ratones que poseían un TI estéril.
Durante la primer colonización identificaron un nuevo fenotipo pequeño y granular SG , diferente del fenotipo liso y grande (LS) de la cepa silvestre. ( Ver figura asociada numero 1). Se puede ver que la cepa es presenta mucha menor movilidad.

Utilizando un biblioteca genómica de la cepa silvestre identificaron que las mutaciones que originaba el fenotipo se encontraban exclusivamente en el locus ompB que codifica para el regulador de dos componentes ompR y el sensor de membrana EnvZ. Este EnvZ/OmpR regula la expresión géníca en respuesta a los cambios en la osmolaridad del medio. En ensayos de competencia utlizando la cepa LS y la cepa SG observaron que la cepa SG ( mutada en envZ) se encuentra favorecida en la colonización respecto de la cepa silvestre. (se puede observar en la coinfección en distintas proporciones: 1:1, rombos, 1:100, cuadrados, 1:1000, triángulos. (Figura 1)
Las mutaciones en envZ o ompR llevan a la disminución en la expresión de fliC. (esto la va gustar a Alfonso) Estos flacos tomaron heces ( si si si caca) de los ratones colonizados y observaron que la expresión de la proteína YFP bajo el promotor de fliC disminuye su fluorescencia a lo largo de los días de colonización. (Observar la figura dos, los círculos llenos corresponden a la disminución de la actividad del promotor de fliC y la disminución en la cepa LS. Los cuadrados corresponden a la una cepa ∆fliC permitiendo ver que esta cepa se pierde pero en mucha menor proporción de que la cepa LS)

Las mutaciones en envZ/OmpR llevan a la falta de motilidad, la disminución en la expresión de las porinas ompC y otros genes implicados en la formación de biofilms. Esto muestra que se prefieren las mutaciones adaptativas en los genes envZ/ompr se prefiere a múltiples mutaciones en otros genes a causa de su efecto pleiotrópico en la expresión génica.
Articulo original: Giraud et al. Dissecting the genetic components of adaptation of Escherichia coli to the mouse gut. PLoS genetics (2008) vol. 4 (1) pp. e2
Publicado en Uncategorized
Tuneando la expresión génica

 tamasa comienze a activarse las concentraciones de Amp disminuirán llevando al apagado de la GFP a causa de la disminución de la concentración de aM-pentapéptido por acción de AmpR sobre el promotor.
tamasa comienze a activarse las concentraciones de Amp disminuirán llevando al apagado de la GFP a causa de la disminución de la concentración de aM-pentapéptido por acción de AmpR sobre el promotor.
Publicado en Uncategorized
